Définition d’une souche épidémique et séquençage génomique : du seuil ‘magique’ aux seuils ‘ad hoc’
François Gravey1 et Sylvain Brisse2
Pour la section Épidémiologie et Génomique des Populations de la Société Française de Microbiologie
Affiliations institutionnelles :
1 Université de Caen Normandie, Univ Rouen Normandie, INSERM, Normandie Univ, Dynamicure UMR 1311, CHU Caen, Département des agents infectieux, F-14000, Caen, France
2 Institut Pasteur, Université Paris Cité, Unité Biodiversité et Epidémiologie des Bactéries Pathogènes, Paris, France
*Correspondance : Pr Sylvain Brisse, Institut Pasteur, Unité Biodiversité et Epidémiologie des Bactéries Pathogènes, 25-28 rue du Docteur Roux, F-75724, Paris, France ; +33 1 45 68 83 34; E-mail : sylvain.brisse@pasteur.fr
A partir de quelle distance génétique est-il possible d’exclure deux isolats comme n’étant pas reliés au même processus de dissémination clonale ? Cet article discute de différentes manières d’aborder cette question.
Introduction
Presque 30 ans après la publication des premiers génomes bactériens (Fleischmann et al. 1995; Kunst et al. 1997; Blattner et al. 1997), les technologies de séquençage à haut débit sont utilisées en routine dans les laboratoires de microbiologie. Le ‘déluge de données’ génomiques a non seulement révolutionné la recherche fondamentale, mais aussi ses applications en surveillance épidémiologique et investigation d’épidémies (Loman et Pallen 2015).
Utilisées dans le cadre de l’exploration des épidémies infectieuses, les informations obtenues par analyse des génomes complets des agents pathogènes permettent de détecter l’origine des souches épidémiques. Une des premières applications a été l’analyse de l’épidémie de choléra en Haïti en 2010, tout comme l’étude de l’épidémie Européenne de Escherichia coli O104:H4 en 2011, qui avait causé de plus de 4000 cas et 50 décès (Chin et al. 2011; Rasko et al. 2011). L’analyse rapide des séquences génomiques des souches incriminées a permis de définir les souches bactériennes impliquées et de tracer leurs origines parmi la diversité phylogénétique des souches répertoriées auparavant. De plus, le séquençage génomique permet de caractériser les facteurs de virulence. Enfin, cette approche permet de mener des études rétrospectives qui permettent de comprendre la dissémination de pathogènes, comme en Colombie Britannique, où a été mise en évidence la circulation concomitante de plusieurs lignées de Mycobacterium tuberculosis en lien avec les réseaux de consommation de cocaïne (Gardy Jennifer L. et al. 2011).
Les souches d’une épidémie sont rapidement apparues comme très reconnaissables par l’emploi du séquençage génomique. Au cours de l’année 2009, le service de réanimation néonatale de l’hôpital « the Rosie hospital » situé à Cambridge a été confronté à une épidémie à Staphylococcus aureus résistant à la méticilline (SARM) (Köser et al. 2012). Le séquençage du génome complet a été réalisé en temps réel pour la première fois, permettant l’utilisation sans délai des résultats par les équipes opérationnelles d’hygiène. Au total, 14 isolats de SARM ont été séquencés, dont dix de séquençotype ST22. Après comparaison avec le génome de la souche ‘index’ prise pour référence, sept isolats épidémiques étaient définis, c’est-à-dire appartenant à la souche épidémique. A l’exception d’une souche présentant des anomalies dans les systèmes de réparation de l’ADN ; souche qui présentait des mutations dans le gène mutS (voir ci-dessous), les souches épidémiques avaient une distance génomique comprise entre 3 et 14 polymorphismes nucléotidiques simples (Single Nucleotide Polymorphism, SNP). Ces mêmes génomes étaient distants d’au moins 102 SNPs par rapport à d’autres isolats, alors définis comme sporadiques (Köser et al. 2012). Cette publication est une des premières illustrations d’un principe central : la définition une distance génomique seuil pour faire la distinction entre souches épidémiques et souches sporadiques, considérées comme non-reliées à l’évènement épidémique investigué (Köser et al. 2012).
Ainsi, si le séquençage génomique a largement remplacé les approches de typage moléculaire utilisées jusque-là (Quainoo et al. 2017), un principe des approches antérieures a été retenu : l’utilisation d’un seuil génétique pour circonscrire la « souche épidémique ». De fait, une question centrale demeure : comment interpréter les données pour définir une « souche épidémique » ? Autrement dit, jusqu’à combien de différences génétiques avec le cas index, peut-on associer ou non un isolat à l’épidémie étudiée ?
Dans un article précédent, « Comparaison des séquences génomiques pour la surveillance épidémiologique et l’investigation d’épidémies : 3 approches complémentaires » de la section épidémiologie et génomique des populations, disponible sur site de la SFM, nous avons décrit différentes approches pour la détermination de la distance génomique: (i) l’alignement des données de séquençage contre un génome de référence; (ii) la comparaison des génomes assemblés par la méthode MLST; et (iii) la comparaison du contenu en séquences nucléotidiques de longueur k (« k-mer ») contenues dans les génomes (Jamin et al. 2021). Nous avons aussi discuté précédemment de la nomenclature des souches lors d’un article intitulé « La taxonomie des souches bactériennes à l’heure de la génomique » également disponible sur le site de la SFM.
Ici, nous discutons de la validité de la notion de seuil pour définir une souche épidémique, quelle que soit la méthode de comparaison des génomes choisis.
Le seuil « magique » : un seuil unique, applicable pour toutes les épidémies d’une espèce d’agent pathogène ?
Au cours de certaines investigations, la distinction entre souches épidémiques et sporadiques semble évidente, comme dans l’exemple ci-dessus de l’épidémie de SARM en Angleterre (Köser et al. 2012), apportant un caractère faussement rassurant à cette idée d’un « seuil magique ». La recherche d’un seuil de distance génétique qui permettrait d’exclure les souches non-reliées à l’épidémie (sporadiques) est une approche pragmatique, nécessaire d’un point de vue opérationnel, pour décider de mesures d’hygiène hospitalière ou guider les investigations d’épidémies alimentaires. L’emploi d’un seuil prédéfini applicable largement est ainsi devenu une pratique généralisée en épidémiologie moléculaire. C’est ce que nous appelons ici par simplicité, le « seuil magique ». Elle semble reposer sur l’hypothèse, pas toujours formulée, que les génomes d’une épidémie auraient une tendance intrinsèque à se démarquer des souches non liées épidémiologiquement. Un exemple célèbre de proposition de seuils, et de critères d’interprétation épidémiologiques associés, est celle de Tenover et collègues, appliquée aux données PFGE ; même si les auteurs eux-mêmes prévenaient que les seuils proposés devaient être vus comme des guides plutôt que des règles absolues (Tenover 1985). Dans l’ère génomique, le seuil recherché est un nombre de SNPs ou d’allèles MLST différents. La prise en compte des différences entre espèces bactériennes en termes de taille de génomes, vitesse évolutive et mécanismes de recombinaison différents (Ochman, Elwyn, et Moran 1999; Biek et al. 2015), a toutefois amené à l’idée de définir un seuil pour chaque espèce bactérienne, plutôt qu’un seuil unique applicable à toutes. Un nombre croissant de publications font état de l’utilisation de seuils génomiques, pour la plupart choisis de façon empirique. Une compilation de ces seuils est, par exemple, disponible dans la revue de Schürch et al. en 2018 (Schürch et al. 2018).
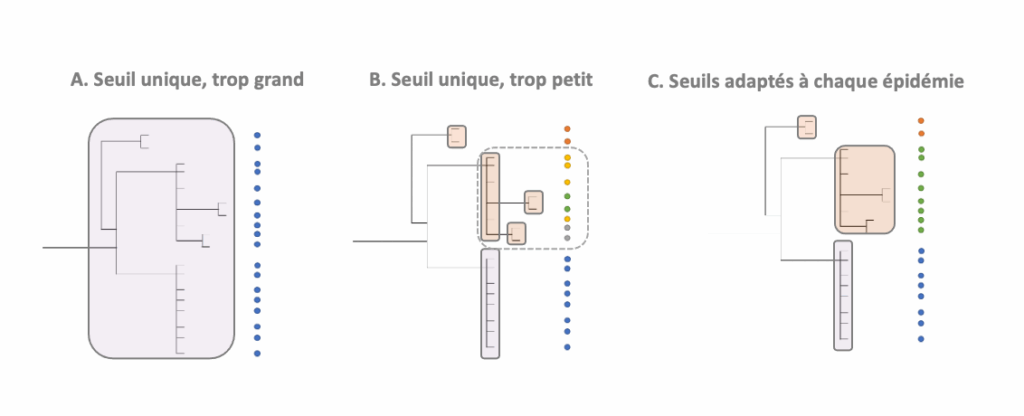
Cependant, l’accumulation d’analyses d’épidémies de la même espèce a montré que selon les cas, il est nécessaire de tolérer plus ou moins de différences pour délimiter la souche épidémique et exclure les sporadiques (Moura et al. 2016; Nadon et al. 2017; Schürch et al. 2018). Et cela s’explique très logiquement, puisque les épidémies peuvent durer plus ou moins longtemps, accumulant ainsi de la variation entre isolats microbiens d’une même épidémie. Par ailleurs, des souches très similaires à la souche épidémique mais n’ayant pas de lien avec l’épidémie, pourraient exister et être échantillonnées, rendant difficile l’exclusion des isolats sporadiques sans aussi exclure des souches épidémiques. Ces simples considérations démontrent qu’il est illogique « d’inscrire dans le marbre » un seuil de distance génomique validant ou éliminant de façon certaine une diffusion clonale, même en s’adaptant espèce par espèce. Autrement dit, il ne peut exister, en théorie, de « seuil magique ». Les effets indésirables de l’application de seuils magiques sur l’exploration d’une diffusion bactérienne sont illustrés dans la Figure 1.
Définir un seuil pour chaque épidémie par une approche de biologie de l’évolution
Mais alors, quel seuil utiliser et comment le définir, puisqu’il en faut bien un ? S’il faut avoir une approche par épidémie, définissant le seuil le plus utile et le plus probable pour le cas particulier de l’épidémie investiguée, on peut craindre un raisonnement circulaire : on définit le seuil par comparaison des isolats sporadiques et épidémiques, eux-mêmes parfois définis en prenant en compte un seuil précédemment utilisé pour une autre épidémie causée par la même espèce bactérienne.
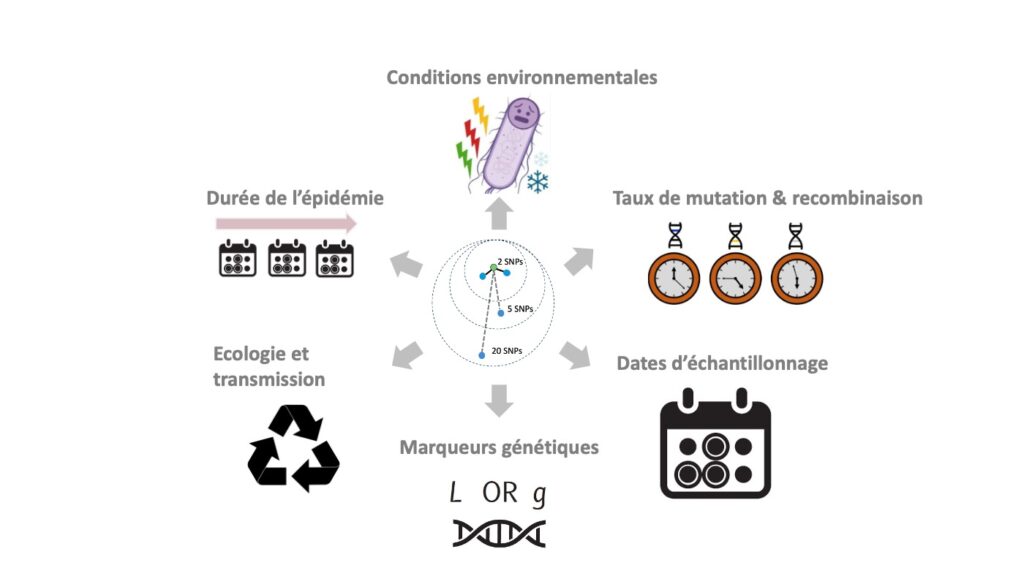
C’est ici qu’une approche de biologie évolutive peut aider, en sortant de ce cercle vicieux. Durant une épidémie, plusieurs facteurs vont influer l’accumulation de différences entre les souches impliquées : la durée de l’épidémie bien sûr, mais aussi le taux de mutation de la souche en question, lui-même pouvant être influencé par les conditions environnementales (stress, vitesse de croissance). Par ailleurs, le nombre de différences observées dépendra des zones du génome bactérien analysées pour les chercher : génome entier, ou sous-ensemble de marqueurs prédéfinis, comme le « core génome » (ensemble de gènes présents dans toutes les souches de l’espèce, généralement beaucoup plus petit que le génome entier) dans l’approche MLST (voir l’article précédent « Comparaison des séquences génomiques pour la surveillance épidémiologique et l’investigation d’épidémies : 3 approches complémentaires ». La recombinaison homologue est de plus bien connue pour accélérer l’évolution chez certaines bactéries (Didelot et al. 2016). Ces paramètres biologiques et épidémiologiques, s’ils étaient connus, pourraient permettre de définir un nombre de différences génétiques attendues entre souches épidémiques, en fonction du temps et d’un modèle d’évolution. Alors, quels sont ces paramètres ? La figure 2 illustre certains d’entre eux.
En premier lieu, il est important de considérer l’impact du temps sur les distances observées entre souches d’une même épidémie. Une publication s’est intéressée à la dissémination de souches de Klebsiella pneumoniae ST258 porteurs du gène de la carbapénèmase blaKPC au Presbyterian Hospital (Université de Pittsburgh) entre 2010 et 2018 (Marsh et al. 2019). Les analyses génomiques ont révélé l’existence de deux clades différents, distants de 364 SNPs, produisant les carbapénèmases KPC-2 ou KPC-3. Au sein des deux groupes, la diversité était, elle aussi, différente (64 SNPs vs. 16 SNPs). Enfin, des sous-groupes ont également été définis sur la base de distances allant de 1 à 27 SNPs (Marsh et al. 2019). Une approche bayésienne s’appuyant sur les distances génomiques observées et les dates d’infection par chaque souche, a déterminé le taux de mutations, permettant d’en déduire la séparation des deux groupes autour de l’année 1996. Cette durée explique le grand nombre de SNPs séparant les isolats de chacune des deux « épidémies » (Marsh et al. 2019).
Deuxièmement, les taux de mutations varient entre souches d’une même espèce. Par exemple, des souches bactériennes dites mutatrices (ayant des taux de mutations beaucoup plus élevés que la moyenne de l’espèce) ont été décrites chez les espèces bactériennes pathogènes, pouvant entraîner une évolution rapide et des distances exceptionnelles (voir l’exemple du SARM cité ci-dessus) (Köser et al. 2012; Denamur et Matic 2006). Au cours de l’exploration européenne de E. coli O104 :H4, quatre souches isolées en Allemagne n’étaient distantes que de deux SNPs, tandis que onze souches isolées en France présentaient une distance de 19 SNPs (bien que cinq isolats retrouvés au sein d’un même individu ne présentaient aucune différence génomique) (Grad et al. 2012). Les phénotypes hyper-mutateurs sont fréquemment associés à des anomalies de systèmes de réparation de l’ADN, notamment les systèmes de mismatch repair impliquant les gènes mutS, mutH, mutL et mutY. Leur disruption entraine des taux de mutations augmentés dans certains cas jusqu’à 150 fois, et ne sont pas rares car ils peuvent générer des mutations avantageuses, au moins transitoirement (Denamur et Matic 2006; Wielgoss et al. 2013).
Troisièmement, les environnements dans lesquels se trouvent les bactéries peuvent influencer les taux de mutation. Par exemple, la congélation va empêcher l’évolution d’une souche épidémique dans une source contaminée, tandis qu’une conservation à 4°C est propice à la croissance de certaines bactéries, comme Listeria monocytogenes qui pourra continuer d’accumuler des mutations génomiques, augmentant la distance mesurée lors des analyses des souches contaminantes. Bien sûr, la mauvaise utilisation de la chaîne du froid favorise la multiplication des bactéries, un problème bien connu dans les contaminations groupées à Salmonella spp. ou Escherichia coli. Enfin, certains stress exogènes favorisent les phénotypes hyper-mutateurs et par conséquents impactent les distances mesurées entre les génomes. Ces stress sont notamment le système immunitaire de l’hôte, la pression antibiotique ou encore certains sites anatomiques. Il a été démontré que le milieu urinaire favorisait la présence des phénotypes hyper-mutateurs à la fois dans des modèles in vitro, mais aussi in vivo dans les phases chroniques d’infections urinaires causées par E coli (Denamur et Matic 2006; Labat et al. 2005). Par ailleurs, les traitements par fluoroquinolones entrainent des mutations dans l’opéron ramAR responsables de la surexpression de la pompe d’efflux AcrAB-TolC. Cette même surexpression a pour conséquence l’inhibition de l’expression du gène mutS alors à l’origine d’un phénotype hyper mutateur (Bialek-Davenet et al. 2011; El Meouche et Dunlop 2018).
Enfin, les modes de transmission et l’écologie des bactéries sont à prendre en compte. Dans le cadre d’investigation d’épidémie interhumaine à Staphylococcus aureus, une étude s’est intéressée aux distances génomiques retrouvées au sein de 20 épidémies distinctes (Gordon et al. 2017). Si la distance génomique médiane des souches épidémiques entre elles était de 13,8 SNPs, contre une distance de 4444 SNPs vis-à-vis de souches non épidémiques, les sept souches les plus divergentes provenaient d’épidémies ayant duré plus de six mois (Gordon et al. 2017). Dans de telles épidémies, les auteurs soulignent l’impact des patients porteurs asymptomatiques de S. aureus sur les seuils épidémiques qu’ils proposaient (distance de 0 à 10 SNPs en l’absence de porteurs chroniques vs. 0 à 46 SNPs). Le portage au long cours est une source de diversification génétique intra-hôte (Didelot et al. 2016), qui devient source de diversité inter-hôtes dans un contexte d’épidémie de cas cliniques observés.
Mais alors comment faire en pratique ? Les approches de modélisation
Ayant à l’esprit les phénomènes indiqués ci-dessus, il apparait fondamental de tenir compte des mécanismes évolutifs à l’œuvre durant une épidémie. Différents modèles évolutifs ont récemment été proposés, prenant en compte les paramètres principaux influençant le seuil génétique tolérable, en particulier la durée de l’épidémie et le taux de mutations des agents pathogènes. Par exemple, une étude portant sur la transmission inter-humaine de Staphylococcus aureus, a proposé de tenir compte de la diversité intra-hôte et les dates de transmission pour définir des évènements directs de transmission (Coll et al. 2020).
A notre connaissance, Octavia et ses collaborateurs en Australie (Octavia et al. 2015) ont été les premiers à tenter de modéliser la variation génétique s’accumulant durant une contamination bactérienne alimentaire. En s’appuyant sur la théorie de la coalescence, et en étudiant cinq épidémies causées par des souches de Salmonella enterica, ces auteurs ont proposé un seuil maximal de divergence de 2 à 4 SNPs pour des souches isolées jusqu’à un mois d’intervalle. Toutefois, bien que les auteurs aient incorporé le taux de mutation et la durée de l’épidémie dans leur modèle, les dates d’échantillonnage (ou de contamination des patients) ne sont pas considérées.
Une modélisation de la distribution des distances génétiques attendues, étant donné (i) une durée de l’épidémie, (ii) des dates de prélèvement et (iii) une vitesse évolutive, a été proposée plus récemment pour définir le seuil génétique applicable (Duval, Opatowski, et Brisse 2023). Cette étude a utilisé le modèle d’évolution des populations de Wright-Fisher, en incorporant les dates d’échantillonnage, qui peuvent avoir un effet important sur les distances génétiques entre souches. Tant dans des simulations que sur des jeux de données réels, cette approche de modélisation classe correctement la plupart des souches épidémiques, excluant ainsi efficacement les souches sporadiques non reliées à l’évènement considéré. Cette étude souligne aussi l’impact de la durée de l’épidémie et taux de mutations sur l’efficacité avec laquelle les génomes sont correctement classés : les prédictions seront plus fiables pour des durées plus étendues et des taux d’évolution plus rapides. Enfin ce modèle, qui utilise des chaines de Markov cachées, peut également être utilisé pour estimer la durée de l’épidémie, ou le taux de mutation de l’agent pathogène (Duval, Opatowski, et Brisse 2023) .
Il est important de souligner que les modèles évolutifs utilisés ont des limites, et en particulier en termes de domaine d’applicabilité. En effet, les mécanismes d’évolution d’une population bactérienne et l’accumulation de la variation génétique en son sein, sont très différents lors d’une contamination alimentaire ou environnementale, par rapport à une chaîne de transmission inter-individus durant laquelle seuls quelques variants, s’il en existe chez le contaminateur, prendront pied chez le nouvel hôte, entraînant un effet de goulot d’étranglement sur la diversité génétique des souches, qui sera donc réduite lors de la transmission interhumaine. En revanche, la population contaminant une source alimentaire pourra évoluer sans que la taille de la population change drastiquement. Ainsi, tester la validité des modèles mathématiques en vie réelle, par exemple dans l’environnement hospitalier, reste un chantier largement inachevé.
Conclusions
Cet article souligne l’importance d’abandonner l’idée d’un seuil ‘magique’ qui serait universellement applicable aux épidémies causées par un agent pathogène donné, au profit d’approches évolutives tenant compte de la situation particulière de chaque épidémie. En effet, pour interpréter en termes épidémiologiques les résultats de séquençage génomique, plusieurs paramètres clefs comme la durée de l’épidémie, le taux de mutation, et les dates d’échantillonnage doivent être considérés. D’autres paramètres entrent en compte, comme la représentativité génomique des marqueurs utilisés, ou la qualité des données étudiées (une mauvaise qualité revient à introduire de fausse mutations). Aussi, à la question récurrente du seuil génétique à considérer pour définir une souche épidémique, la réponse sera au cas par cas. Après plus de 15 ans de développement de l’épidémiologie génomique, l’idée d’un seuil magique a la vie dure. Si les développements des approches de modélisation sont un pas dans la bonne direction, il reste à mettre en place des outils pragmatiques pour faciliter leur utilisation par les microbiologistes, épidémiologistes et hygiénistes, et à répertorier les taux de mutation les plus probables pour chaque agent pathogène.
Remerciements
Les auteurs remercient Maxime Pichon pour ses commentaires lors de la préparation de cet article.
Déclaration de licence
Pour l’intérêt de l’accès libre, les auteurs déclarent avoir appliqué une licence publique de droit d’auteur CC-BY pour toute version auteur du manuscrit résultant de cette soumission.
Références bibliographiques